bregr 是一个专为生物医学研究设计的开源 R 软件包,致力于简化回归分析中的批量建模流程。该工具融合了 tidyverse 的数据处理风格与 S7 面向对象编程架构,支持多种回归模型的高效自动化分析,包括广义线性模型、Cox 比例风险模型以及混合效应模型。通过原生 R 管道操作符(|>)实现链式调用,结合内置的并行计算机制和稳健的错误处理系统,bregr 显著提升了大规模数据集的处理效率与分析可重复性。
该软件包已在 CRAN 正式发布,并在 TCGA 数据集上进行了实际验证,展现出良好的性能扩展能力与实用性,为生物医学领域的研究人员提供了一套结构清晰、操作便捷的回归建模解决方案。
核心亮点
- 多模型支持:涵盖线性回归、逻辑回归、Cox 回归等多种常用统计模型。
- 统一管道流程:完全兼容原生 R 管道语法,支持函数链式调用,提升代码可读性与可维护性。
- 集成可视化功能:一键生成森林图、残差诊断图等高质量图表,满足科研发表需求。
- 整洁结果输出:返回结果采用 tidyverse 兼容格式,便于后续整合与进一步分析。
- 批量自动化处理:支持对多个因变量或自变量进行快速循环建模,显著减少重复编码工作。
引言
回归分析作为生物医学统计的核心方法之一,广泛应用于探索临床结局与各类预测因子(如分子标志物、临床指标等)之间的关联。在实际研究中,科研人员常需构建大量单变量或多变量回归模型以筛选关键变量,传统方式往往依赖逐一手动建模,过程繁琐且易出错。尽管部分研究者尝试编写脚本来实现批量处理,但这类自定义脚本通常缺乏标准化输出、可重复性差,并难以应对复杂数据结构。
现有工具在结果组织、可视化输出及高维数据处理方面仍存在明显短板,亟需一个功能全面、稳定高效的自动化解决方案。在此背景下,我们团队此前开发了专注于生存分析的 ezcox 包,虽仅以预印本形式发布,但已获得超过 40,000 次下载,反映出社区对自动化回归工具的强烈需求。基于这一经验,我们进一步推出 bregr —— 一款面向更广泛回归任务的 R 工具包。
bregr 不仅继承了用户友好的设计理念,还引入了更强的可扩展性与稳定性。其采用 S7 类系统构建,确保模块化与可拓展;遵循 tidyverse 哲学,使数据流处理更加直观流畅。同时,集成并行计算能力使其能够高效处理包含数千样本与变量的大规模组学数据集,已在 TCGA 多癌种队列中成功应用,验证了其在真实场景下的可靠性与高性能。
01 软件包实现与分析流程概述
1.2 软件包实现
bregr 的设计围绕“简洁、一致、可扩展”三大原则展开。其底层基于 R 的 S7 面向对象系统,将不同类型的回归模型封装为统一接口的对象类,允许用户通过一致的方式调用不同模型。所有核心函数均支持原生 R 管道操作,无需中间赋值即可完成从数据输入到结果输出再到可视化的完整流程。
为了提高执行效率,bregr 内置并行计算模块,可通过简单参数设置启用多核加速,尤其适用于高维基因表达数据或大规模临床数据库的批量建模任务。此外,系统具备完善的错误捕获与警告提示机制,在遇到共线性、缺失值过多或模型不收敛等问题时,能自动跳过异常模型并保留有效结果,保障整体流程的连续性。
1.1 数据集集成
为全面展示 bregr 的功能特性,我们在多个真实与模拟数据集上进行了测试与演示。其中包括 R 自带的 mtcars 和 survival 包中的 lung 数据集,用于基础功能验证。
为进一步体现其在生物医学场景中的适用性,我们整合了 TCGA-LUAD(肺腺癌)队列中 513 名患者的临床信息,并联合 IOBR R 包提供的 258 个癌症相关特征。这些特征通过 ssGSEA 方法计算得出,覆盖肿瘤微环境、代谢通路及肿瘤内在亚型等多个维度。所有 ssGSEA 得分直接作为输入传入 bregr,未经过额外转换或标准化处理,充分体现了该包对原始数据的鲁棒性支持。
为评估其在高维数据下的性能表现,我们还采用了 TCGA-BRCA(乳腺浸润性癌)队列的基因表达矩阵,包含超过 1210 个样本和 20,000 余个基因,已预处理为适合批量 Cox 回归分析的格式。
基准测试所用数据集由 10,000 行观测和 1,001 列变量构成,使用以下函数模拟生成:
set.seed(2025L)rnorm()该模拟数据集的设计旨在模拟真实高通量组学实验中的数据规模与结构,可用于复现完整的性能评估流程,相关代码可通过公开网站获取。
02 软件包设计思路与核心功能
2.1 设计理念
bregr 软件包基于 tidyverse 风格构建,旨在简化 R 语言中的批量回归建模流程(见图 1)。该工具采用 S7 面向对象编程框架,用于创建可扩展的批量回归对象,支持同时执行多个单变量与多变量回归模型。用户能够定义多个自变量,并在短时间内高效运行数百个模型。
其核心特性包括:以整洁格式输出结果、集成多种可视化工具(如森林图和风险网络图),以及通过原生 R 管道操作符(|>)实现连贯的数据分析流程。这些设计确保了与 R 生态系统的无缝对接,显著增强了数据分析与结果呈现的能力。
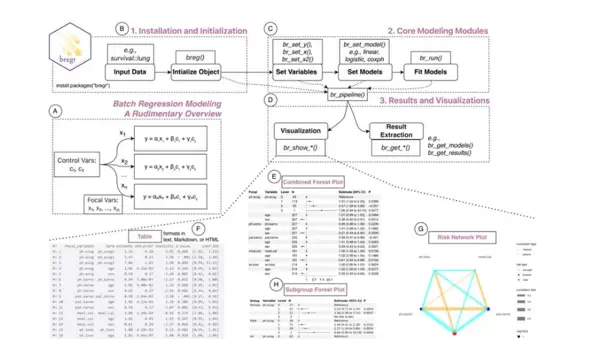
图1. 肿瘤微环境中的外泌体信号网络
(A) 批量回归建模过程的整体概览。bregr 的工作流程涵盖:(B) 安装与初始化,(C) 核心建模执行,(D) 结果提取及可视化。具体可视化功能包括:(E) 森林图,(F) 批量生成的回归结果表格,(G) Cox 回归的风险网络图,(H) 针对固定自变量的分组分析变体森林图,以及通过 (I) 比较分析或 (J) 单至双变量回归函数进行的模型诊断。其中,图 (E-G) 基于 lung 数据集生成,图 (I-J) 使用 mtcars 数据集完成。
2.5 支持多种回归类型
bregr 兼容多种常用回归模型,涵盖广义线性模型(例如线性回归、逻辑回归)以及 Cox 比例风险回归,充分满足生物医学研究中常见的统计建模需求。用户可根据实际场景自定义模型参数(如观测权重),提升方法在不同研究背景下的适应性和灵活性。
2.2 批量处理能力
该软件包通过允许用户明确指定因变量、自变量、可选的控制变量以及目标回归类型(如线性、逻辑或 Cox 回归),实现了自动化拟合所有变量组合对应的回归模型。这一机制特别适用于快速评估变量影响、依据统计指标筛选最优模型,或在共享控制条件下重复建模,从而大幅减少编码负担。
此外,bregr 支持引入分组变量(如性别、临床分期等)进行分层建模,提升了在不同生物医学子群体中探索预测因子效应差异的灵活性与实用性。
2.3 结构化输出与结果提取
bregr 提供
br_get_*()2.4 集成可视化
bregr 内置强大的可视化功能,有效提升结果解读效率。例如,森林图清晰展示各模型的效应估计及其置信区间,便于跨模型比较;而专为 Cox 回归设计的风险网络图,则提供直观的方式揭示变量间的关联模式与风险结构。
这些图形工具不仅增强结果的可读性,也支持研究人员更有效地传达复杂建模发现,尤其适用于科研论文、项目汇报等正式场景。
1.3 回归建模分析工作流程
使用 bregr 进行批量回归建模的标准流程包含以下关键步骤:
- 初始化批量回归对象:通过
创建初始对象。breg() - 设置变量:利用
定义因变量,br_set_y()
指定自变量,以及br_set_x()
引入可选的控制变量。br_set_x2() - 选择模型类型:使用
设定回归模型种类(如 "gaussian" 表示线性回归,"coxph" 对应 Cox 回归)。br_set_model() - 执行模型拟合:调用
函数,高效完成所有预设模型的拟合任务。br_run() - 提取并可视化结果:通过
提取建模结果,并结合br_get_*()
等函数生成高质量图表。br_show_forest()
1.4 一步式管道操作
为了进一步简化操作流程,bregr 提供了
br_pipeline()结论
bregr 软件包通过融合高效的批量建模能力、整洁规范的结果输出、丰富的可视化选项以及模块化的函数架构,显著优化了 R 中的大规模回归分析体验。它使研究人员能够在较短时间内构建、比较并解释大量回归模型,极大提升了工作效率。
作为一款新发布的工具,bregr 仍需在更多实际应用场景中接受验证,以持续改进其稳定性和适用范围。尽管存在一定的局限性,但其已在 R 生态系统中展现出强大潜力,成为生物医学领域开展高效、可重复回归分析的重要补充。
bregr 可从 CRAN 和 GitHub 获取,开发版本可通过 R-universe 安装。GitHub 平台提供完整的文档资源,包括类与函数参考手册、入门示例及高级应用指南,并配备议题追踪系统以支持社区协作与问题反馈。




 雷达卡
雷达卡




 京公网安备 11010802022788号
京公网安备 11010802022788号







