摘要
多组学研究旨在揭示不同实验条件(如对照组与处理组)下分子生物学机制的动态变化。这一目标通常依赖于分析多种生物分子——包括脂质、代谢物、蛋白质等基因产物——之间的相互作用或功能关联的变化。虽然已有多个公共数据库收录了部分物种中分子间的关系信息,但对于研究较少的非模式生物,这些资源往往极度匮乏,因此必须借助科学文献来补充数据来源。
从文献中提取此类关系面临诸多挑战,例如:需要自动化处理全文内容、整合生物术语的多种同义表达形式,以及部署复杂的自然语言处理和机器学习模型。为降低技术门槛并提升可访问性,研究人员开发了一个名为 DancePartner 的 Python 软件包。该工具能够从科研文献和现有数据库中高效提取生物分子间的功能关系,支持将不同命名的同义词映射至标准标识符,并具备构建与可视化多组学网络的能力。
本文展示了两个示例数据集的应用效果:其一涵盖 14,986 篇与秀丽隐杆线虫(Caenorhabditis elegans)相关的论文及摘要;其二包含 33,606 篇关于酿酒酵母(Saccharomyces cerevisiae)的研究资料。所提取的关系进一步整合自 KEGG、WikiPathways、UniProt 和 LipidMaps 等权威数据库,并通过网络图谱进行可视化呈现。同时,研究还比较了不同方法在构建这些网络时所需的时间开销。
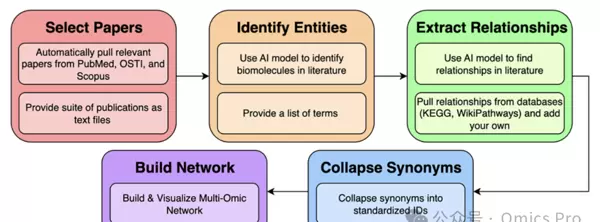
实施流程概述
DancePartner 提供了一套模块化的分析流程,用户可根据需求灵活选择各步骤中的具体方法:
- 输入文献可通过程序自动从指定数据库拉取,也可由用户提供本地文本文件;
- 目标实体识别阶段,既可使用内置的 ScispaCy 工具进行生物命名实体识别(NER),也可直接导入预定义的实体列表;
- 关系提取支持两种路径:基于文献挖掘的关系抽取,或从已知数据库中获取注释关系,且两者可组合使用。
完成上述流程后,系统会自动将识别出的生物分子同义词归并到统一的标准标识符下,最终生成可用于下游分析的多组学关系网络。
安装与资源链接
项目源码托管于 GitHub:
https://github.com/pnnl-predictive-phenomics/DancePartner
预训练模型地址(Hugging Face):
https://huggingface.co/david-degnan/BioBERT-RE
Scopus API 接口文档:
https://dev.elsevier.com/
ScispaCy 自然语言处理工具:
https://allenai.github.io/scispacy/
配套教程手册(共4篇):
https://github.com/PNNL-Predictive-Phenomics/DancePartner/tree/main/vignettes
图1:DancePartner 分析流程示意图
每个主要步骤(大框)包含若干可选操作(小框),用户可根据实际需求自由组合配置。
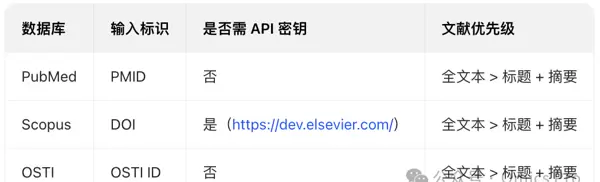
拉取文献(Select Papers)
支持的数据源与输入格式如图所示。
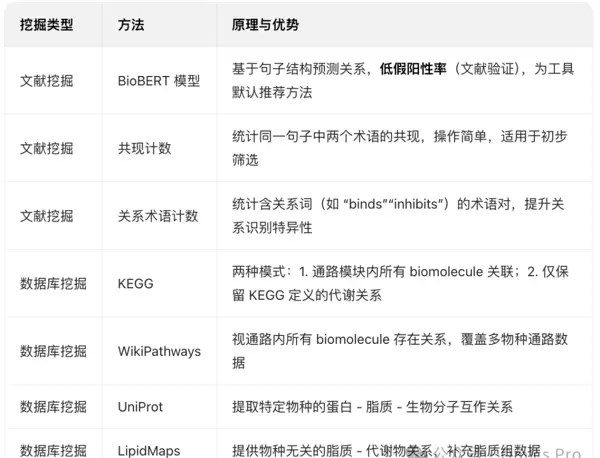
提取关系(Extract Relationships)
分为“文献挖掘”与“数据库挖掘”两大类别,支持联合使用以增强覆盖范围。
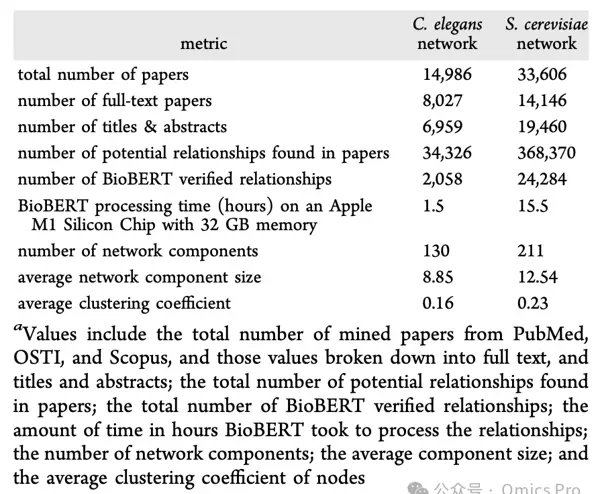
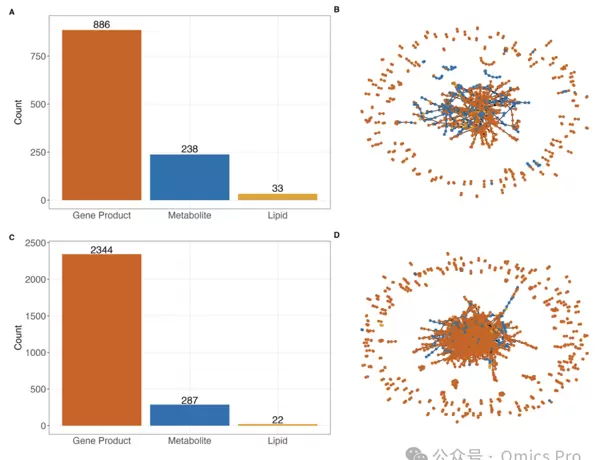
表1:秀丽隐杆线虫与酿酒酵母网络指标对比
利用 DancePartner 构建的两个模式生物多组学网络的关键统计指标对比如下。
图2:多组学网络构建结果
(A)从 14,986 篇秀丽隐杆线虫相关文献中检测到、并与至少一种其他分子存在关系的生物分子总数;
(B)基于 A 中文献构建的秀丽隐杆线虫多组学网络结构;
(C)从 33,606 篇酿酒酵母相关文献中识别出的、具有至少一个关联的生物分子数量;
(D)结合文献与数据库信息构建的酿酒酵母多组学网络(基于 C 中术语筛选)。其中,朱红色节点表示基因产物,蓝色代表代谢物,橙色代表脂质;边表示经文献或数据库确认的两术语间的关系。
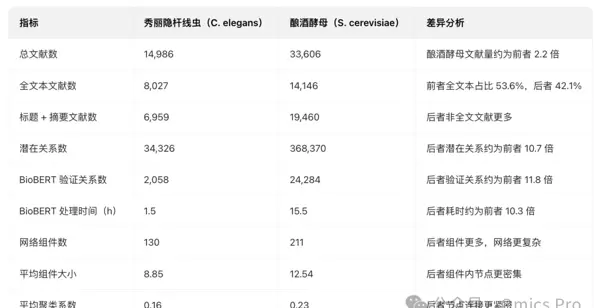
性能验证结果
在 Apple M1 芯片(配备 32 GB 内存)环境下,使用两个模式物种数据集对 DancePartner 进行性能测试,关键构建时间对比如图所示。

核心方法比较:BioBERT 模型 vs 数据库挖掘
DancePartner 提供的两种主要关系提取方式——基于 BioBERT 的文献挖掘与基于数据库的关系提取——各自具有不同的优势与局限性,图中列出了推荐使用场景与逻辑依据。
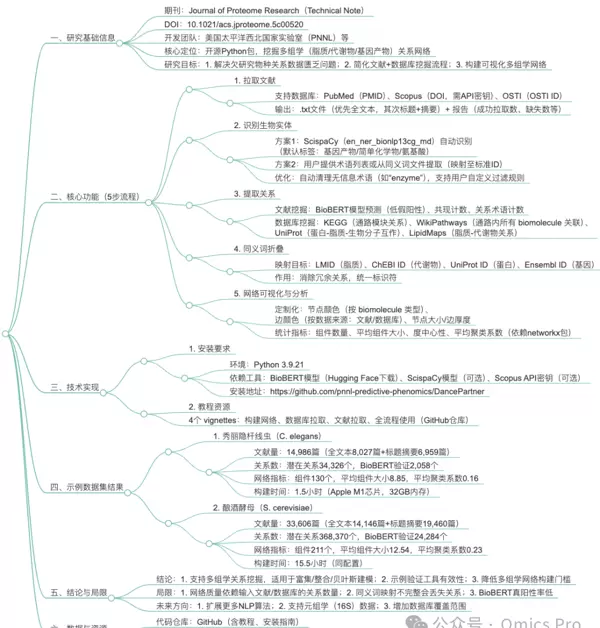
思维导图:整体分析框架概览
参考文献
J Proteome Res. 2025 Nov 4. doi: 10.1021/acs.jproteome.5c00520. DancePartner: Python Package to Mine Multiomics Relationship Networks from Literature and Databases




 雷达卡
雷达卡




 京公网安备 11010802022788号
京公网安备 11010802022788号







