之前给大家写过一篇数据清洗的文章,解决的问题是你拿到原始数据后如何快速地对数据进行处理,处理到你基本上可以拿来分析的地步,其中介绍了如何选变量如何筛选个案,变量重新编码,如何去重,如何替换缺失值,如何计算变量等等------R数据分析:数据清洗的思路和核心函数介绍
今天呢,就更进一步,对于一个处理好的数据,我们就可以进行统计分析了,本文的思路就是对照期刊论文的一般流程写写如何快速的实现一篇论文的统计过程并简洁高效地展示结果。依然提醒大家,请先收藏本文再往下读哈。
先做描述统计基本上文章结果部分一上来首先展示的就是描述统计,就是你有多少样本,样本特征是啥样的----连续变量的均值标准差是多少,分类变量的频数百分比是多少等等,这些都是描述统计
做法3:使用tableone包
做描述统计第三个方法就是用tableone包,依然是对于图1中的数据,我现在想做一个描述统计,连续变量用均值±标准差,分类变量用频数百分比表示,我就可以写出如下代码:
(tab_nhanes <- CreateTableOne(data = data))运行后得到如下描述统计结果:
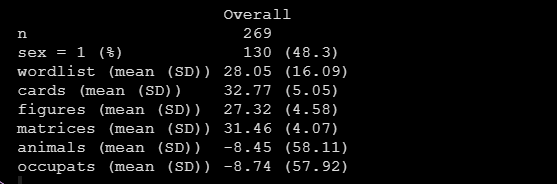
可以看到,sex变量是用频数百分比进行描述的,其余的连续变量都是以均值标准差呈现的。
在使用tableone包的时候如果你通过正态性检验发现某个变量不是正态分布的,这个时候需要用中位数和四分位数间距进行描述,此时在打印tableone对象的时候加上nonnormal = "变量名"参数就好了,比如我现在知道我的数据中年龄是不服从正态分布的,我就可以写出如下代码:
print(tab_nhanes, showAllLevels = TRUE, nonnormal = "Age" )大家肯定见过这样的表格展示的描述统计,就是分组描述统计:
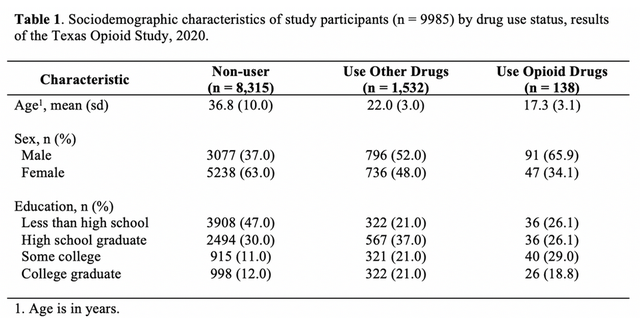
比如干预实验中对照组和干预组的特征比较,两组随访数据的基线特征比较等等。
这样的表格用tableone也是非常容易实现的,比如我的原始数据长这样:
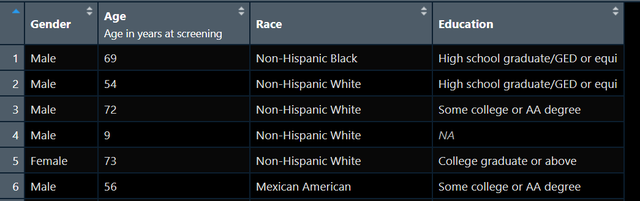
图2
我现在想以Gender这个变量进行分组描述统计,我便可以写出如下代码:
strata <- CreateTableOne(data = data, vars = c("Age", "Race", "Education"), factorVars = c("Race","Education"), strata = "Gender")print(strata, nonnormal = "Age", cramVars = "Gender")上面的代码中,strata参数设置分组变量,factorVars指定变量类型为因子,vars参数指定我们要进行统计描述的变量,运行后出来的结果如下:
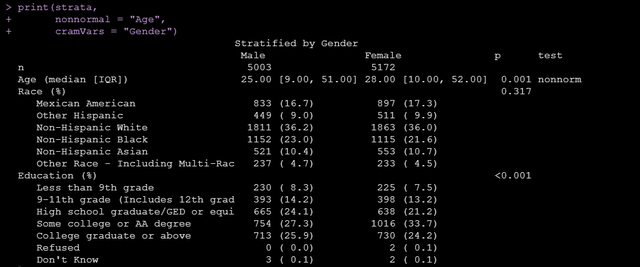
可以看到既有所有变量的统计描述还有组间比较的p值,另外我们可以很方便地通过以下代码将做出来的tableone输出成csv:
tab_csv <- print(strata, nonnormal = "Age", printToggle = FALSE)write.csv(tab_csv, file = "Summary.csv")运行后即可在目录中找到相应的csv文件,然后直接复制粘贴到论文中。
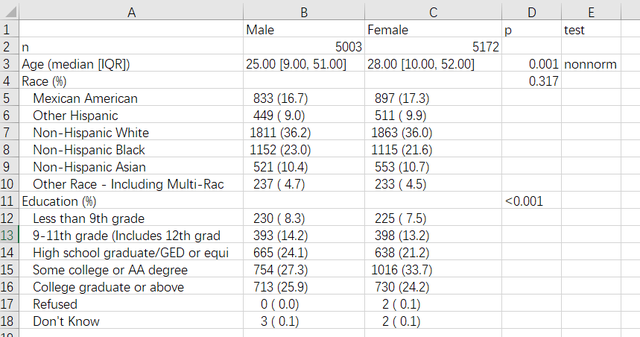
图3
方法4:gtsummary
最后要给大家介绍的方法就是使用gtsummary中的tbl_summary函数,比如依然是上面的数据(图1中的数据),我使用gtsummary函数写出代码如下:
data %>% tbl_summary( by=Gender,statistic = all_continuous() ~ "{mean} ({sd})",) %>% add_p() %>% modify_caption("**Table 1. Please follow Wechat Channel--Codewar**")可以看到,代码基本就1行,add_p是添加分组比较的p值(按需使用),modify_caption是更改表的标题,运行上面的代码,即可得到又一张出版级的表格如下(内容和图3也是一样的):
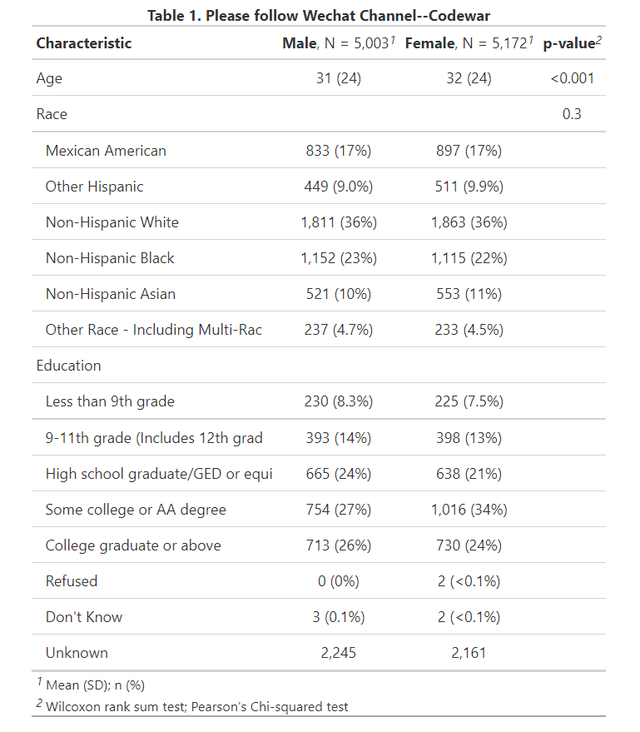
图3
真棒!这个表格也可以通过write.csv输出为excel然后直接贴到你的论文中。
再做相关分析描述统计做完了之后我们有可能会需要做一下各个变量间的两两相关,期刊中常见的比较标准的相关结果表示方法如下,变量均值和标准差占两列,然后相关矩阵放后面:
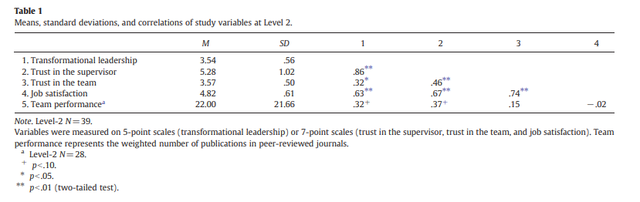
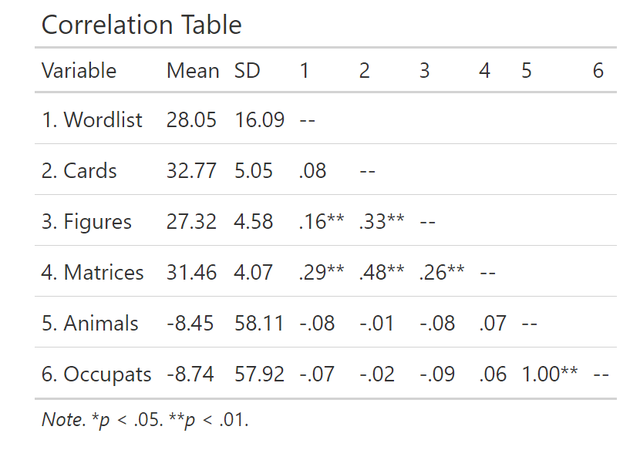
这样的表格也有十分简单的做法,大家可以直接使用mlmCorrs这个包,比如对于图1中的数据,我想拉一个和上图一样布局的结果表格,我只需要直接运行下面的代码:
data %>% select(wordlist:occupats) %>% mlmCorrs::corstars()便可以得到结果如下,真的是很方便呀:
再做主分析
变量间的相关关系做完之后,大家要做多因素分析了,比如你要做个多元线性回归,比如你要做个逻辑斯蒂回归,或者做个生存分析,这些分析是你论文中最重要的部分,也是你的主要研究结论的体现。
这儿也给大家展示几个例子,首先写个简单的多元线性回归,其余的直接改相应的主分析函数就行。
方法1:tab_model
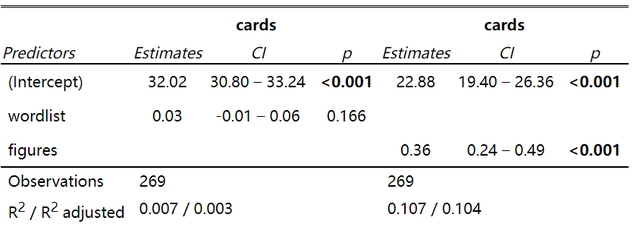
依然是图1中的数据,我现在随意跑了两个线性回归模型,代码如下:
model1 <- lm(cards ~ wordlist, data=data_txt)model2 <- lm(cards ~ figures, data=data_txt)我想要展示模型的信息,只需要运行下面的代码就可以:
sjPlot::tab_model(model1, model2)得到的结果:
方法2:gtsummary
刚刚写了线性回归的例子,再给大家看看logistics回归和cox回归的模型展示,我先用同一批数据拟合一个logistics模型和一个cox模型:
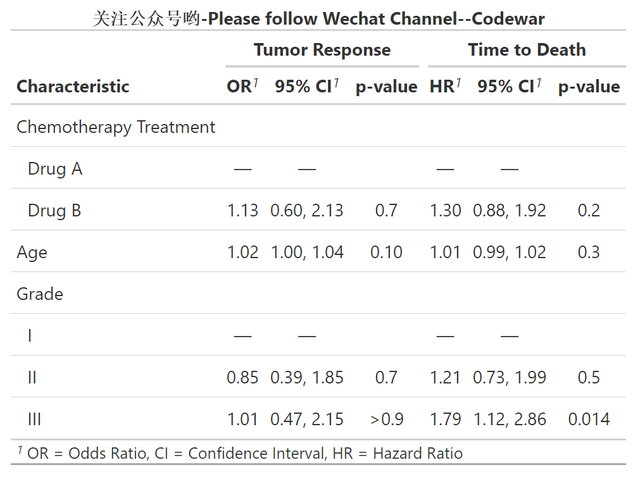
glm(response ~ trt + age + grade, trial, family = binomial) %>% tbl_regression(exponentiate = TRUE) coxph(Surv(ttdeath, death) ~ trt + grade + age, trial) %>% tbl_regression(exponentiate = TRUE)此时我可以用tbl_merge函数将两个模型合并起来展示(这也是多个模型时的常规展示方法),代码如下:
tbl_merge_ex1 <- tbl_merge( tbls = list(t1, t2), tab_spanner = c("**Tumor Response**", "**Time to Death**") ) %>% modify_caption("**关注公众号哟-Please follow Wechat Channel--Codewar**")运行后输出结果如下:




 雷达卡
雷达卡




 京公网安备 11010802022788号
京公网安备 11010802022788号







