AI重磅突破!H&E图像秒变多重免疫荧光,肿瘤微环境建模迈入新纪元!
关键词:多模态AI、肿瘤微环境、虚拟多重免疫荧光、H&E图像、临床发现
本研究提出了一种名为GigaTIME的创新性多模态人工智能框架,旨在实现对肿瘤免疫微环境(TIME)的大规模、高精度建模。该框架基于深度学习技术,能够将常规获取的苏木精-伊红(H&E)染色切片转化为包含21个蛋白质通道的高保真虚拟多重免疫荧光(mIF)图像,从而在无需额外实验成本的前提下,重建出空间分辨的蛋白质组学信息。
GigaTIME的训练依托于一个大规模配对数据集,涵盖超过4000万个细胞的H&E与真实mIF图像,使其具备了从细胞形态推断复杂蛋白表达模式的能力。研究人员将该模型应用于来自51家医院及1000多家诊所的真实世界癌症患者队列,共涉及14,256名个体,成功生成近30万张虚拟mIF切片。这一规模远超传统mIF技术可及范围,显著缓解了因数据稀缺和检测昂贵带来的研究瓶颈。
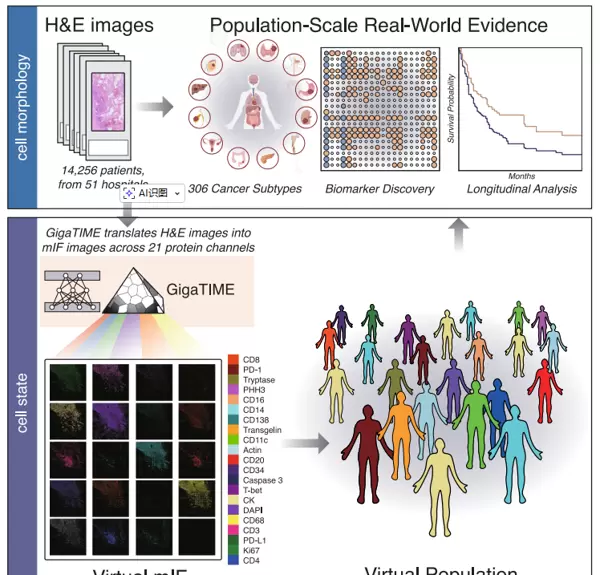
通过分析这些虚拟生成的数据,研究团队识别出1,234个具有统计学意义的关联,涵盖了蛋白质表达、生物标志物状态、病理分期以及患者生存结果等多个维度。这不仅揭示了多种新型空间共激活模式,还为患者分层和预后预测提供了新的依据。此外,在包含10,200名TCGA患者的独立队列中进行的验证表明,GigaTIME的预测结果具有良好的泛化能力和稳健性。
该研究展示了人工智能在精准免疫肿瘤学中的巨大转化潜力。通过将广泛存在的H&E图像转化为富含空间信息的虚拟蛋白质图谱,GigaTIME为深入理解肿瘤与免疫系统的相互作用机制开辟了新路径,有望推动癌症诊断策略和治疗方案的革新。
研究亮点
- 利用多模态AI技术,将常规H&E病理切片转换为高保真的空间蛋白质组学数据。
- 构建基于H&E图像的“虚拟人群”,支持超大规模肿瘤微环境分析。
- 突破传统mIF技术的数据稀缺与高成本限制,赋能大规模临床发现与个体化分层。
- 揭示前所未有的空间分布特征与组合蛋白激活模式,深化对肿瘤免疫互作的理解。
局限性说明
- 当前研究样本主要来源于美国西部地区,未来需拓展至更多元化的地理与种族背景以提升代表性。
- 部分蛋白质难以仅凭组织形态准确还原,其表达可能无法完全由H&E图像推断。
- 翻译质量存在波动,可能受组织异质性、阳性事件频率差异及标记物特异性等技术因素影响。
破解大脑密码!AI驱动非编码变异功能解读,神经发育疾病研究新突破!
关键词:非编码变异、功能基因组学、神经干细胞、增强子活性、BRAIN-MAGNET
一项前沿研究开发了BRAIN-MAGNET——一种全新的卷积神经网络,并配套构建了一个全面的功能基因组学图谱,用于系统解析非编码遗传变异在神经发育过程中的作用。研究采用ChIP-STARR-seq技术,在人类神经干细胞(NSCs)中对148,198个非编码调控元件(NCREs)进行了功能性注释,为理解大脑发育的基因调控机制提供了关键资源。
该图谱不仅揭示了NSCs中复杂的调控网络,还首次发现:许多在胚胎干细胞阶段的非编码调控元件已处于“预先激活”状态,为后续神经分化和功能建立做好准备。这一发现挑战了传统线性调控观念,提示早期发育阶段即存在远期神经活动的调控预设。
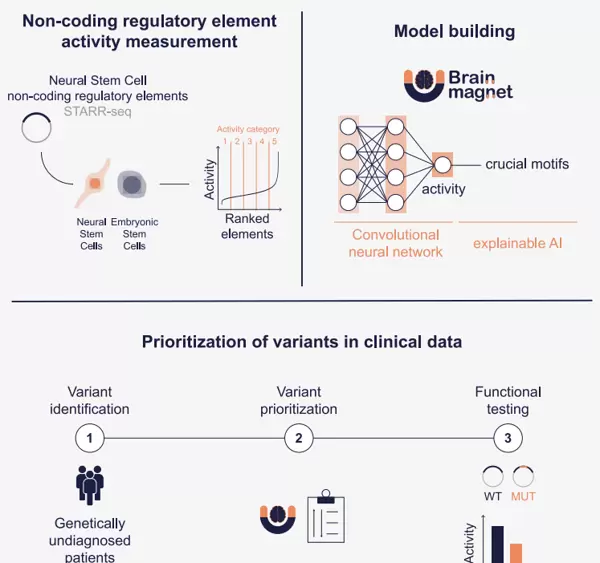
基于上述图谱,BRAIN-MAGNET模型被训练用于直接从DNA序列预测NCRE的增强子活性,并精准识别维持其功能所必需的关键核苷酸位点。该工具在全基因组关联研究(GWAS)位点的精细定位中表现出卓越性能,能有效优先筛选出与常见神经系统疾病相关的非编码变异,同时助力识别病因未明的罕见神经遗传病患者的候选致病变异。
通过一系列实验验证,包括NCRE缺失和单碱基点突变测试,证实了BRAIN-MAGNET在预测功能效应方面的高准确性,且整体表现优于现有主流变异预测工具。这项成果为非编码基因组的功能注释提供了强有力的新方法,尤其在发现潜在增强子病变方面具有重要价值。
研究亮点
- 建立了包含148,198个神经干细胞调控区域的功能基因组图谱,奠定非编码区研究基础。
- 发现胚胎干细胞中部分非编码元件已被预先激活,为后期神经发育提供调控储备。
- BRAIN-MAGNET可从序列水平预测增强子活性,并锁定关键功能核苷酸。
- 有效识别与常见及罕见神经系统疾病相关的关键非编码变异,促进疾病机制解析与诊断。
局限性说明
- MPRA实验在游离染色质环境中检测NCRE活性,可能未能完全模拟体内内源性染色质的真实状态。
- 活性量化缺乏统一内参对照,可能导致跨实验比较时出现偏差。
研究揭示了长臂猿基因组的演化历程,通过整合现存与已灭绝物种的基因组数据,全面解析了其系统发育关系、种群历史及关键形态特征的遗传基础。该研究利用染色体水平的参考基因组、18种现存长臂猿的全基因组重测序数据以及3个古代样本的线粒体基因组信息,构建了一个高分辨率的进化图谱。
全基因组比对分析明确了长臂猿四个属之间的系统发育关系,确立了(Hylobates, (Nomascus, (Symphalangus, Hoolock)))这一分支模式,解决了长期以来在分类学上的争议。值得注意的是,通过对古线粒体DNA的分析,原本被归为独立属的君子长臂猿(Junzi imperialis)被重新归入Nomascus属,修正了此前的分类认知。
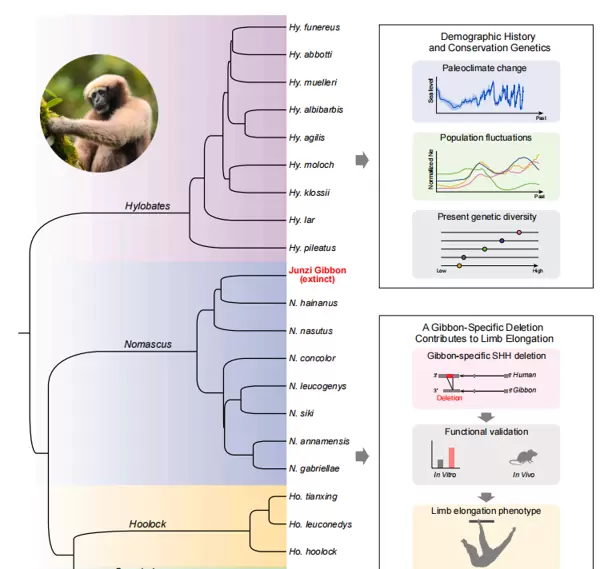
保护基因组学与生态位模型联合分析显示,气候变化显著影响了长臂猿种群规模和栖息地适宜性的历史动态。这些发现为当前受威胁长臂猿物种的保护策略提供了重要的历史背景支持。此外,比较基因组学结合转基因小鼠实验鉴定出Sonic Hedgehog (SHH)基因调控区域中一个特异性的205bp缺失,该结构变异与长臂猿独特的肢体伸长表型密切相关。
尽管取得了重要进展,但研究仍存在一定局限性:目前尚未获得两种现存长臂猿(N. nasutus 和 Hy. albibarbis)的核基因组数据,且海南长臂猿缺乏高覆盖度基因组序列,可能遗漏部分关键进化信号。古DNA分析仅基于三个样本的线粒体基因组,只能反映母系遗传信息,限制了对整体种群历史的深入推断。同时,每个物种取样数量有限,难以精确重建近期尤其是人类活动加剧时期的种群变化过程。此外,纯合性片段(RoHs)分析虽有助于评估近交程度,但不足以单独预测灭绝风险,因其受多种因素共同作用。
本研究不仅深化了对长臂猿系统发育、生物学特性及保护现状的理解,也为未来灵长类动物的进化机制与保护基因组学研究提供了坚实的数据基础和方法范式。
另一项突破性研究则从永久冻土中成功恢复并测序了晚更新世猛犸象的古老RNA(aRNA),组织样本保存时间介于1万至5万年前之间。其中,约3.9万年前的Yuka个体提供了高质量的转录组数据,首次实现了对已灭绝物种组织特异性基因表达的系统解析,特别是在骨骼肌代谢通路中观察到活跃的调控特征。
这是迄今为止记录到的最古老的RNA序列,标志着古遗传学研究从DNA时代迈向转录组层面的新阶段。研究还利用aRNA表达证据,支持了猛犸象基因组中多个新型微小RNA候选位点的功能注释,提升了基因组功能元件识别的准确性。
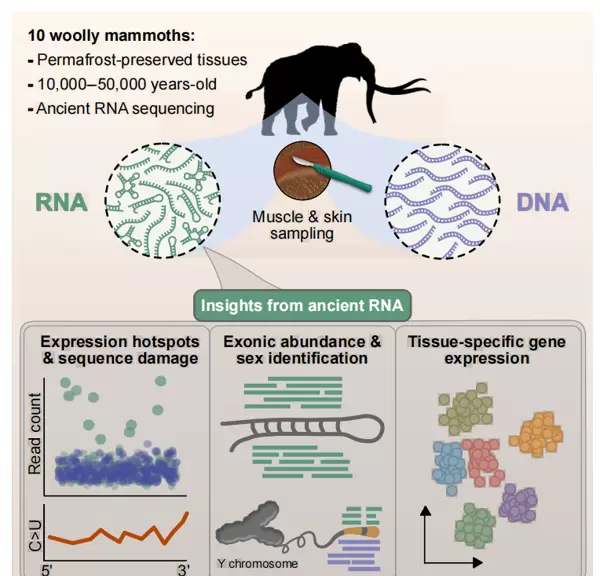
该工作建立了一套完整的aRNA提取、测序、分析与验证方法体系,证明了跨越数万年的RNA分子仍可保留生物学相关信息。结果表明,组织特异性表达模式能够在化石材料中长期保存,从而揭示仅靠基因组DNA无法获取的生命活动细节。这为理解灭绝物种的生理状态、发育调控和适应机制打开了全新窗口。
然而,当前研究仍面临若干挑战:所分析样本局限于冻土保存的软组织(如骨骼与皮肤),尚需探索矿化组织中RNA的保存潜力;现有aRNA数据覆盖度较低,亟需更多样本以构建全面的古转录组图谱;RNA分离与建库技术尚未完全优化,可能导致部分分子丢失;尽管已采取严格质控措施,仍不能完全排除外源DNA污染对结果的潜在干扰。
总体而言,这两项研究分别在基因组与转录组层面上拓展了我们对已灭绝物种的认知边界,展示了多组学整合在古生物学研究中的巨大潜力,预示着一个融合基因组学、蛋白质组学与转录组学的综合古生物研究新时代的到来。
细胞感染病毒并非随机!揭示细胞内在状态如何决定病毒易感性!
关键词:单细胞、病毒感染、细胞状态、SARS-CoV-2、流感病毒A
研究发现,细胞对病毒的易感性并非完全由其类型决定,而是受到细胞自身所处功能状态的深刻影响。并非所有具备受体表达的细胞都会被病毒感染,真正起决定作用的是细胞在特定时间点所处的动态状态。通过应用一种名为“Rewind”的单细胞克隆追踪技术,研究人员成功回溯了人类Calu-3细胞中哪些子群体更容易被SARS-CoV-2感染,并识别出两类高度易感的状态:一类表现为TIG1基因高表达,另一类则处于快速增殖阶段。
为了验证这些状态的功能重要性,研究团队对TIG1高表达状态下显著上调的多个因子(包括AXL、TSPAN8、TIG1和MUC20)进行了敲除实验。结果显示,这些基因的缺失显著降低了病毒感染效率,提示它们可能在病毒进入过程或维持可感染性细胞状态方面发挥关键调控作用。
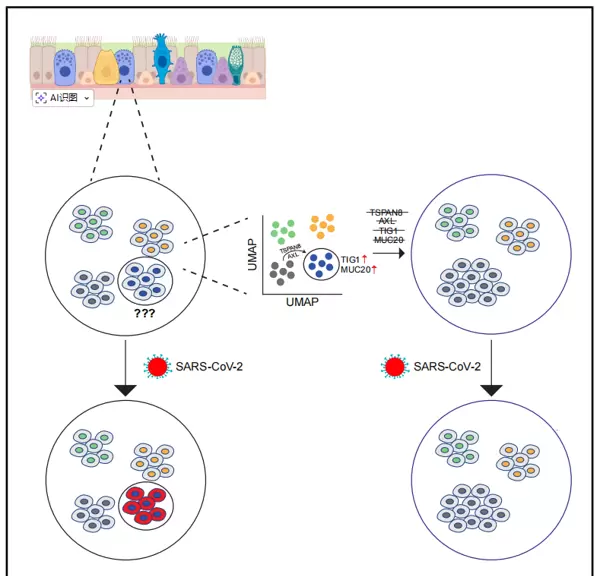
进一步分析表明,不同病毒倾向于感染具有不同分子特征的细胞状态。例如,以KRT8与CD46高表达为标志的一类细胞状态,则对流感病毒A表现出更高的易感性,说明病毒感染的选择性具有病毒特异性。这一发现强调了宿主细胞状态与病原体之间的精准匹配关系。
值得注意的是,这些在体外鉴定出的易感细胞状态,在真实的人体肺组织样本中也被检测到存在,并且与炎症相关病理变化显著关联,暗示其在临床疾病进展中可能扮演重要角色。该研究为理解病毒感染异质性的根本机制提供了新的视角,同时也为开发针对特定易感细胞状态的抗病毒干预策略开辟了全新路径。
研究亮点:
- 细胞对病毒的易感性主要取决于其内在功能状态,而非简单的细胞分类。
- 成功鉴定出对SARS-CoV-2和流感病毒A分别高度易感的不同细胞亚群,体现病毒特异性选择。
- 识别出的易感细胞状态在人体肺部组织中真实存在,并与炎症性疾病相关,具备潜在临床价值。
- 通过基因编辑手段证实AXL、TSPAN8、TIG1和MUC20等因子在病毒感染中的功能性作用。
研究局限性:
- 目前关于体内易感细胞状态与疾病严重程度之间的联系仍停留在相关性层面,缺乏因果证据支持。
- 研究主要依赖于体外细胞模型,尽管已有部分体内数据验证,但仍需更多动物模型及时间序列分析来深入阐明其生物学意义。
- 病毒感染本身会迅速改变宿主细胞的转录图谱,因此在感染发生前准确捕捉易感细胞的真实状态存在技术挑战。
创新疗法!IgE致敏肥大细胞精准递送溶瘤病毒,激活抗肿瘤免疫!
关键词:肥大细胞、IgE、靶向递送、溶瘤病毒、癌症免疫疗法
本研究开发了一种新型的细胞介导药物递送平台,利用经过工程化处理的免疫球蛋白E(IgE)致敏肥大细胞(IgE-MCs),实现对肿瘤部位的靶向输送并增强抗癌免疫应答。该系统巧妙地将肿瘤特异性抗原作为“假过敏原”,引导IgE-MCs在抗原阳性肿瘤区域富集。当遇到相应抗原时,这些细胞被激活并发生脱颗粒反应,释放出预先封装的溶瘤腺病毒(OVs)、趋化因子以及多种促炎介质,从而有效重塑免疫抑制性的肿瘤微环境。
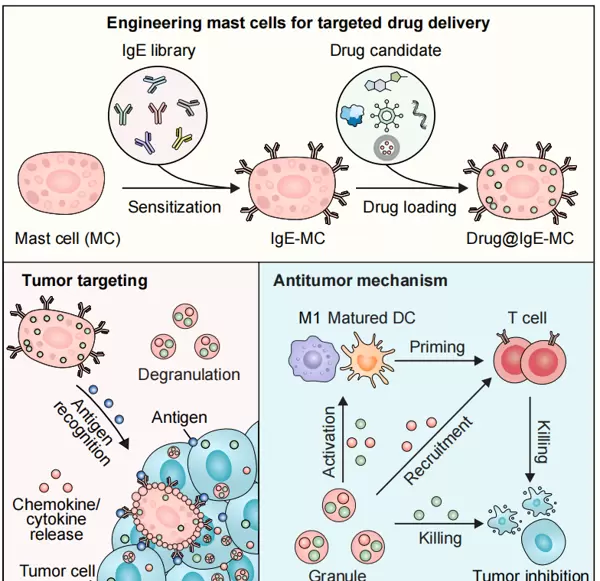
实验证明,装载了溶瘤病毒的IgE-MCs在多种小鼠肿瘤模型中均展现出强大的治疗效果,包括皮下移植瘤、肺部转移灶以及免疫抑制性强的4T1乳腺癌模型。治疗后不仅显著抑制了肿瘤生长,还诱导了持久的抗肿瘤免疫记忆。
在人源化HER2阳性患者来源异种移植(PDX)模型中,使用携带抗HER2 IgE并负载OVs的人源IgE-MCs进行干预,同样观察到瘤内T细胞浸润明显增加、肿瘤体积减小,且未引发血管生成或转移倾向等副作用。安全性评估显示,输入的IgE-MCs可在约两周内被机体自然清除,未诱发全身性过敏反应,也未在正常组织中检测到病毒复制现象。
该平台具有高度模块化特性,可根据患者个体情况选择特异性IgE,并兼容多种治疗载荷(如不同类型的溶瘤病毒或其他免疫调节剂),展现出在个性化肿瘤治疗中的广阔前景。
研究亮点:
- IgE致敏的肥大细胞可作为靶向工具,特异性聚集于表达相应抗原的肿瘤部位。
- 该递送系统具备模块化设计优势,支持个体化IgE筛选与多样化治疗药物封装。
- 抗原触发可激活IgE-MCs释放溶瘤病毒及免疫刺激分子,实现局部高效响应。
- 能够逆转肿瘤免疫抑制状态,激活内源性T细胞反应,提升整体抗肿瘤疗效。
研究局限性:
- 有效抗原的选择是当前主要瓶颈,肿瘤异质性和抗原逃逸现象增加了靶点筛选难度。
- 类似于CAR-T疗法,肥大细胞的遗传改造流程复杂且成本较高,可能制约其大规模临床推广。





 雷达卡
雷达卡




 京公网安备 11010802022788号
京公网安备 11010802022788号







