摘要:掌握Linux命令行操作是进行生物信息分析的关键基础。本文系统整理了在生物信息学研究中必须熟练使用的Linux核心命令,并结合实际应用场景详细说明其用途,帮助初学者快速入门并高效处理测序数据、基因组文件和注释信息等。
一、核心理念:以数据处理为导向的Linux使用观
不必对命令行感到畏惧。您的目标并非成为系统管理员,而是学会利用这些工具高效地处理生物数据,如FASTQ、GTF、SAM等文本格式文件。建议采用以下学习路径:
了解有哪些可用工具 → 明确何时使用它们 → 掌握如何组合多个命令完成复杂任务
文末将推荐一个实用的在线资源网站,便于随时查阅命令细节。
二、第一阶段:基础导航与文件管理——生存必备技能
这是登录远程服务器后最先需要掌握的操作集合,确保您能自如地浏览目录结构并管理文件。
1. pwd(Print Working Directory)
功能说明:显示当前所在目录的完整路径。
典型场景:当在深层目录中迷失位置时,使用该命令迅速确认当前位置。
2. ls(List)
功能说明:列出当前目录下的所有文件和子目录。
常用选项:
-l以长列表形式展示(包含权限、所有者、大小、修改时间)
-h与上述选项配合,使文件大小以K、M、G等人类可读单位显示
-a显示包括隐藏文件在内的所有内容(隐藏文件通常以
.开头)
示例命令:
ls -lh /home/user/data/ # 查看数据目录的详细内容3. cd(Change Directory)
功能说明:切换当前工作目录。
关键用法:
cd ~cdcd ..cd -4. mkdir / rm(创建/删除目录)
功能说明:用于新建空目录或删除空目录。
重要参数:
mkdir -p使用示例:
mkdir -p project/{raw_data,scripts,results}5. cp / mv / rm(复制 / 移动 / 删除)
功能说明:分别实现文件或目录的复制、移动(也可用于重命名)、删除操作。
极度重要提醒:Linux下删除操作不可恢复!不存在“回收站”机制!
安全操作建议:
- 执行
rmlsrm -irm -rrm -rf6. cat / less / head / tail(查看文件内容)
cat:适用于快速查看小文件内容,也常用于合并文件或输出重定向
less(最常用):分页浏览大文件内容,支持上下滚动、搜索(按
/输入关键词查找),退出方式为按
qhead / tail:分别查看文件开头或结尾的若干行,默认显示10行,可通过参数调整
三、第二阶段:文本处理利器——精准操控生物数据
生物信息学中的大多数数据均为纯文本格式(如FASTA、FASTQ、GTF、CSV等),以下命令将成为您处理这些数据的核心工具。
1. grep(Global Regular Expression Print)
功能说明:在文件中搜索指定字符串或正则表达式模式。
生物信息应用:从注释文件中提取特定基因条目、筛选日志中的错误信息、统计FASTA文件中的序列数量等。
常用选项:
-i-v-c-E-w使用示例:
grep -c "^>" genome.fa # 统计FASTA文件中的序列条数(以>开头的行)
grep -c 'R' plink.fam # 统计plink.fam文件中包含'R'的行数2. awk
功能说明:强大的列式文本分析工具,特别适合处理表格型数据(如制表符或空格分隔的文件)。
基本语法结构:
awk '模式 {动作}' 文件应用场景:提取某一列数据、根据条件过滤记录、执行简单数值计算。
内置变量说明:
$1, $2, ... $NFNR实例演示:
# 打印第1列和第3列,用制表符分隔
awk '{print $1, "\t", $3}' input.txt
# 输出第5列值大于0.05的所有行
awk '$5 > 0.05 {print $0}' differential_genes.txt
# 计算第2列的平均值
awk '{sum+=$2} END {print sum/NR}' data.txt3. sed(Stream Editor)
功能说明:流编辑器,可用于查找替换、删除行、插入文本等自动化文本修改任务。
生物信息应用:批量重命名文件(结合循环脚本)、统一数据格式、字符替换(如标准化染色体命名)。
经典用法:
sed 's/old_string/new_string/g' input.txt > output.txt将文件中所有的
old_string替换为
new_string其中
s表示执行替换操作,
g表示全局替换(每行多次替换)
4. cut / paste / sort / uniq / wc
cut:按列切割文本内容
cut -f 1,3 -d "," file.csv # 用逗号分隔符,取第1,3列sort:对文本行进行排序
-n-kuniq:去除连续重复的行(需先排序);常与sort联用去重:
sort file.txt | uniq -c # 统计每个唯一项的出现次数wc:统计文件的行数、词数和字节数
wc -l huge_file.fastq # 快速知道你有多少条测序序列(行数/4)四、第三阶段:提升效率——软件、权限与进程管理
1. 软件查找与管理(which,包管理工具)
which:查询某个命令或程序的安装路径
which bwa生物信息推荐方案:优先使用 conda(推荐 mamba 加速)来安装和管理软件环境,它能自动解决复杂的依赖关系问题(初学者可暂不深入,后续再逐步掌握)
下载地址参考:
wget -c https://repo.anaconda.com/archive/Anaconda3-2021.05-Linux-x86_64.sh
bash Anaconda3-2021.05-Linux-x86_64.sh2. 文件权限设置(chmod, chown)
chmod:修改文件或目录的访问权限。例如,为脚本添加可执行权限:
chmod +x my_script.sh3. 进程管理(ps、top、kill、nohup、&)
&:在命令末尾添加该符号,可使任务在后台运行,不占用当前终端。
nohup:使用此命令可让进程在用户退出登录后依然持续执行,常用于长时间运行的任务。
nohup command > output.log 2>&1 &ps / top:用于查看系统中正在运行的进程状态及其对CPU、内存等资源的占用情况。其中ps适合查看瞬时状态,top则提供动态实时监控界面。
kill:通过指定进程ID(PID),可以终止某个正在运行的进程,是管理系统负载和异常任务的重要手段。
kill -9 PID # 强制终止4. 输入/输出重定向与管道(>、>>、|)
>:将命令的标准输出结果重定向至指定文件,并覆盖原文件内容。
>>:与“>”类似,但采用追加方式写入文件,不会删除已有内容。
|(管道):【核心功能】将前一个命令的输出作为下一个命令的输入,实现多命令串联操作,极大提升处理效率。
例如在生物信息学分析中,常通过管道组合多个工具进行数据过滤与统计:
zcat reads.fastq.gz | head -n 400000
# 解压、取前40万条序列、进行质控五、学习路径建议
边用边学:无需死记硬背所有命令参数。从实际需求出发,比如:“我想查看测序数据(FASTQ格式)中的前几条序列”,然后逐步查找并应用相关命令。
组合使用:尝试将以下命令通过管道连接起来完成复杂任务:
grepawksortuniq这种组合思维是掌握Linux操作的核心。
善用帮助文档:遇到不熟悉的命令时,可通过以下方式获取帮助信息:
man command例如使用
man grep来查阅详细的官方手册,或使用
command --help以及
-h获得简洁实用的快速参考。
编写Shell脚本:当你需要重复执行一系列相同命令时,就到了学习编写bash脚本的时候了。这是迈向自动化分析流程的第一步,推荐从简单脚本开始练习。
*.sh六、经典综合示例
实现如下功能:统计某基因组文件中包含多少条染色体,并计算每条染色体上标记的数量分布。
# 假设文件是 dp4miss0.2.bim
# 1. 提取染色体
# 2. 计算每行染色体出现次数
# 3. 排序
# 4. 统计每个染色体出现的次数
cut -f 1 dp4miss0.2.bim |sort|uniq -c > chr.list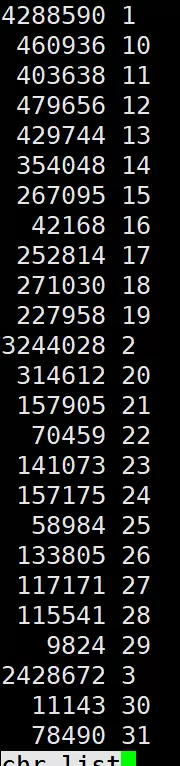
七、实战数据集推荐与练习
针对生物信息学初学者,建议从以下三类基础数据格式入手进行实操训练:
1. FASTA 格式数据(适用于 grep、awk 等命令练习)
数据来源:
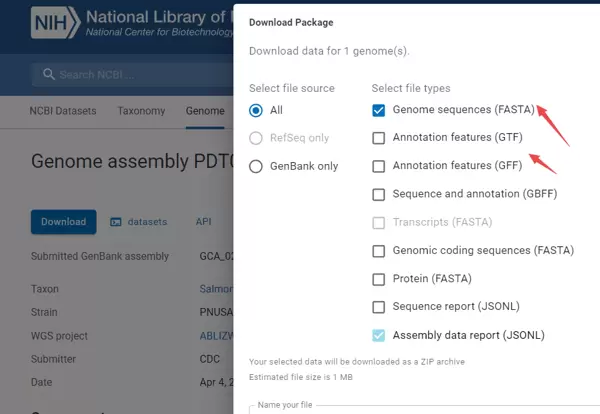
- NCBI 数据集平台:
https://www.ncbi.nlm.nih.gov/datasets/genome/GCA_029584285.1/
- Ensembl Plants:
https://plants.ensembl.org/Setaria_italica/Info/Index
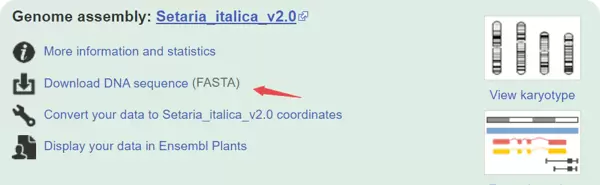
下载方式:支持手动下载或使用脚本自动获取,关键命令为 wget。
练习任务:
grep -c "^>" genome.fa
# 提取所有序列ID
grep "^>" genome.fa | sed 's/>//g'
# 计算总碱基数
grep -v "^>" genome.fa | tr -d '\n' | wc -c
#结果
grep -c "^>" genome.fa
9
grep "^>" genome.fa | sed 's/>//g'
1
2
3
4
5
6
7
8
9
grep -v "^>" genome.fa | tr -d '\n' | wc -c
4224738522. FASTQ 格式数据(适合 awk 和管道操作练习)
数据来源:GEO数据库
访问地址:https://www.ncbi.nlm.nih.gov/geo/
下载流程:利用之前介绍的 wget 命令进行数据抓取。
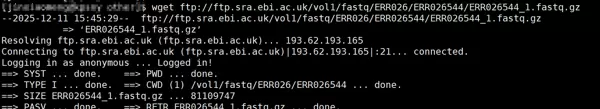
推荐样本:ERR026544(金黄色葡萄球菌的测序数据)
测序平台:Illumina
# 1. 下载文件
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/ERR026/ERR026544/ERR026544_1.fastq.gz
# 2. 查看基本信息
ls -lh ERR026544_1.fastq.gz
# 输出:-rw-r--r-- 1 user group 2.5M ERR026544_1.fastq.gz
# 3. 解压(可选,很多工具可以直接处理gzip压缩文件)
gunzip ERR026544_1.fastq.gz
# 4. 查看文件内容
head -n 4 ERR026544_1.fastq
# 或不解压直接查看
zcat ERR026544_1.fastq.gz | head -n 4
# 5. 统计序列数
echo "序列数: $(( $(wc -l < ERR026544_1.fastq) / 4 ))"
# 或不解压统计
zcat ERR026544_1.fastq.gz | awk 'END{print NR/4}'3. 注释文件(GTF/GFF 格式,适合 cut、sort、uniq 练习)
数据来源:可从 Ensembl 或 UCSC 下载标准基因注释文件(同上文链接)。
练习任务:
# 统计不同特征类型
cut -f 3 annotation.gtf | sort | uniq -c
# 提取特定染色体的注释
grep "^chr1\t" annotation.gtf > chr1_annotation.gtf
# 统计每个基因的外显子数
awk '$3=="exon" {print $10}' annotation.gtf | sort | uniq -c八、常见问题与解决方案
Q1:如何查询某个命令的具体用法?
# 方法1:使用man手册
man grep
# 方法2:查看帮助信息
grep --help
# 或
grep -hQ2:如何查看文件的开头几行或结尾几行内容?
# 查看前5行
head -n 5 file.txt
# 查看后5行
tail -n 5 file.txt
# 同时查看首尾
(head -n 10; tail -n 10) < file.txtQ3:如何查找特定进程并将其终止?
# 终止进程(假设PID为12345)
kill 12345
# 强制终止
kill -9 12345九、总结与进阶建议
最重要的一点是:勤于实践,敢于尝试(请确保在非关键数据环境中操作)。当遇到错误提示时,不要忽略,应仔细阅读报错信息,并将其作为关键词使用搜索引擎进行检索——这是解决技术难题的核心能力之一。
记住,在生物信息学领域,Linux命令行是一个强大的超级工具箱。每一个命令都是一件专用工具,而真正的技巧在于懂得如何将它们灵活组合,以应对具体的科研问题。
祝您在生物信息学的学习道路上不断进步!
提示:本文所列命令示例均已简化,实际应用中需根据具体场景调整参数。建议在虚拟机或测试服务器环境中先行练习,避免在生产系统中造成误操作。
下一期内容预告:
《生物信息学常用数据库完全指南》,将深入讲解 NCBI(美国)旗下的 SRA、GEO 等重要数据库的使用方法。
相关资源:
Linux命令速查表:https://www.runoob.com/linux/linux-command-manual.html




 雷达卡
雷达卡




 京公网安备 11010802022788号
京公网安备 11010802022788号







